Científicos de Estados Unidos crearon un Atlas Metagenómico apoyados en la inteligencia Artificial de Meta: ESMFold.
De acuerdo a publicación de la revista Science, expertos usaron la Inteligencia Artificial de Meta: ESMFold, para levantar un Atlas Metagenómico, el cual dispone de más de 600 millones de proteínas metagenómicas.
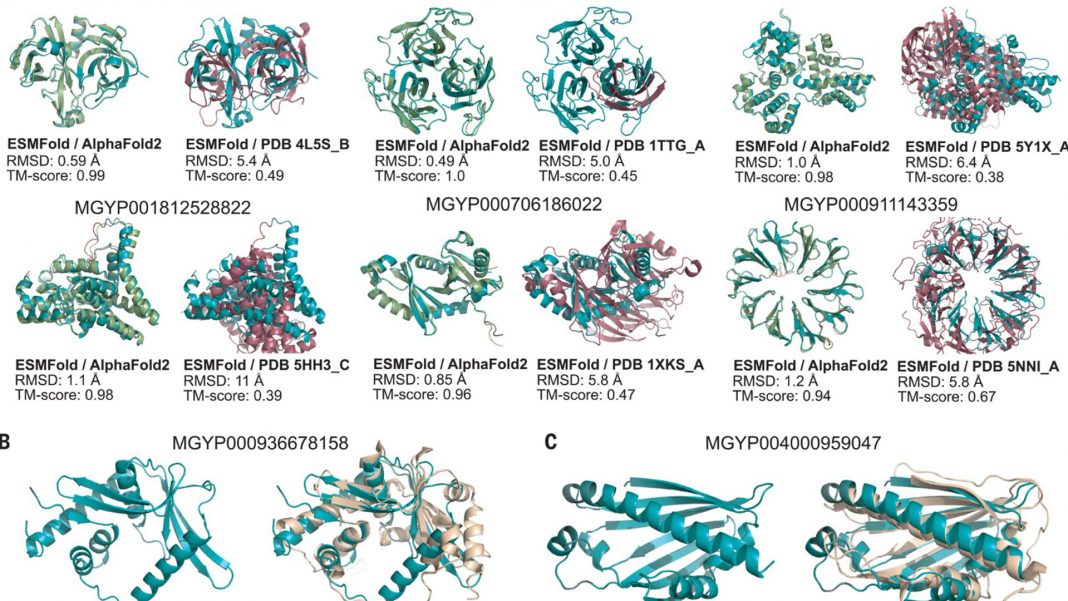
Para ello, se entrenó al ESMFold, el cual se encargó de desarrollar los modelos de lenguaje de proteínas. En este sentido, se transformaron hasta 15 mil millones de parámetros en estructuras predichas experimentales.
ESMFold se dio a conocer en noviembre de 2022, cuenta con una base de datos de 617 millones de proteínas posibles. La misma base dispone de microorganismos, como bacterias y virus, que aún no se han caracterizado.
La intención es mejorar la efectividad de los medicamentos existentes, y ayudar a detectar moléculas que son difíciles de tratar para curar enfermedades como el cáncer, VIH, entre otros.
IA para funciones biológicas
Los avances recientes en el aprendizaje automático han aprovechado la información evolutiva en múltiples alineaciones de secuencias y con ello predecir la estructura de las proteínas.
Por consiguiente, las secuencias de proteínas a la escala de la evolución contienen una imagen de la estructura y función biológicas.
Indican que las propiedades biológicas de una proteína limitan las mutaciones a su secuencia que se seleccionan a través de la evolución. Y con ello poder registrar la biología en patrones evolutivos
Los científicos estadounidenses recalcan que esta capacidad de predicción de estructura rápida y de alta resolución permite la caracterización estructural a gran escala de proteínas metagenómicas.
Los programadores alimentaron esta herramienta con un seriado de letras que representan aminoácidos. Indispensables en la formación del código genético de una proteína, luego ESMFold aprendió a llenar las secciones vacías u ocultas en la secuencia de proteínas.
Toda vez que la secuencia estuvo completa, el programa de Meta acertó la relación. Llegando a determinar entre las secuencias conocidas para predecir las estructuras de las nuevas proteínas.
In a Science study, @MetaAI researchers show the power of a large language model, #ESMFold, to enable protein structure prediction and analysis.
Using ESMFold, they generated a database—the ESM Metagenomic Atlas—of over 600 million metagenomic proteins. https://t.co/jfOUtFy95a pic.twitter.com/wqCnxBWDwx
— Science Magazine (@ScienceMagazine) March 25, 2023